Life Technology二代测序平台
随着生物科学的发展,对模式生物进行基因组重测序以及对一些非模式生物进行全基因组测序已成为较为普遍的研究方法,然而传统的Sanger测序技术已经无法在短期内完成如此数量巨大的测序任务。于是费用更低、通量更高、速度更快的新一代测序技术(Next-generation sequencing),也称为第二代测序技术应运而生。利用新一代测序技术人们能够在更短时间、更低花费的情况下,对基因组序列信息、甲基化修饰调控、RNA表达水平、DNA与蛋白间相互作用等层面进行更为全面、深入地研究分析工作。
佰臻研究院拥有生命科技公司(Life Technology)研发生产的系列测序仪,包括以经典Sanger技术为原理的大型一代DNA测序仪Applied Biosystems 3730XL,还有以SOLiD技术为原理的大型二代测序仪SOLiD 4和SOLiD 5500XL,以及在2010年新推出的小型测序仪Ion Torrent。
(一) Ion Torrent 测序系统
1.检测原理
Ion Torrent(图2-5), 即个人化操作基因组测序仪(简称PGM™),其革命性的设计之处是结合了半导体芯片技术,使整个平台具有极高的扩展性和快速测序的性能。该技术使用了一种布满小孔的高密度半导体芯片,每个小孔就是一个测序反应池。当试剂通过集成的流体通路进入Ion Torrent半导体芯片中,密布于芯片上的反应孔立即成为上百万个微反应体系。DNA聚合酶每把一个核苷酸聚合到延伸中的DNA链上时,都会释放出一个氢离子,反应池中的pH值也会随之发生改变,此时位于池下的离子感受器感受到信号,把化学信号转化为数字信号,从而读出DNA序列。与其它的新一代测序仪相比,它不需要激发光、CCD成像仪或荧光标记,就能直接快速“读”出DNA序列。
Ion Torrent 测序仪通过专有的大规模并行半导体感应器,对于DNA 扩增时产生的离子流,实现直接和实时的检测。这种独特的流体体系、微体系机械设计和半导体的技术组合,使得该系统得以快速直接地将遗传信息翻译成DNA序列信息,从而在短时间内获得大量的测序数据(图2-6)。
.jpg)
图2-5. Ion Torrent 个人化操作基因组测序仪
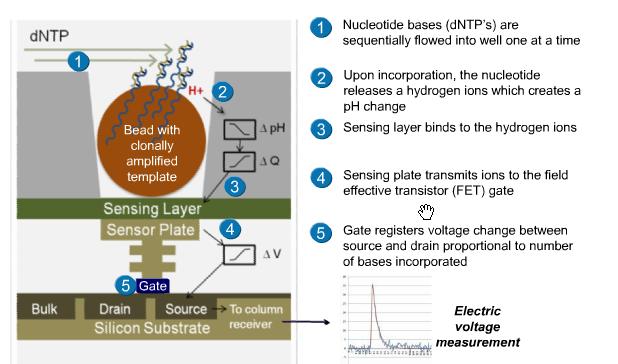
图2-6. Ion Torrent测序原理及基本流程
2.技术特点
(1) 灵活性:大规模可扩展的半导体芯片技术,比拟半导体技术升级的强大扩展性,可满足不同通量的测序要求。
(2) 技术简洁:基于半导体技术测序原理,无需光学部件;简单的生物化学原理,实时的离子流检测过程,无修饰的核酸,无需酶化学级联反应,无需任何荧光、化学发光;且对样本量要求更低。
(3) 快速:从文库构建到数据产出只需2天,上机测序2小时,初步数据分析1小时。
3.应用领域
Ion Torrent测序平台是中等规模测序项目的最佳选择。目前主要应用于:
(1) 小基因组测序(如:微生物和病毒的从头测序和重测序;线粒体测序等)。
(2) 扩增子重测序(如:16S宏基因组测序)。
(3) 靶向重测序。
(4) 基因组/全外显子验证。
4.样品要求
(1) 基因组DNA:5-10μg纯化后DNA。
(2) RNA:不少于10μg的纯化后RNA。
(3) 小RNA:不少于10μg的纯化后RNA。
(4) 外显子捕获:不少于5μg的基因组DNA。
(二) SOLiD 4/SOLiD 5500测序系统
1.基本原理
SOLiD全称为Sequencing by Oligonucleotide Ligation and Detection,它的独特之处在于以四色荧光标记寡核苷酸进行连续连接合成为基础,取代了传统的聚合酶连接反应,可对单拷贝DNA片段进行大规模扩增和高通量并行测序,并能明显减少因碱基错配而出现的错误,消除相位不同步的问题,获得更高的保真度。
SOLiD系统的基本工作流程大体如下:
(1) 文库构建
SOLiD系统能支持两种测序模板:片段文库(fragment library)或配对末端文库(matepa-ired library)。片段文库就是将基因组DNA打断,两端连接测序接头,制成文库,适用于转录组测序、RNA定量测序、miRNA测序、已知基因组重测序、3’,5’-RACE测序、甲基化测序分析、ChIP测序等。如果需要进行全基因组测序、SNP分析、结构重排/拷贝数变异,则需要用配对末端文库。配对末端文库是将基因组DNA打断后,与中间接头连接,再环化,然后用EcoP15酶切,使中间接头两端各有27bp的碱基,再在两端连接上测序接头,形成文库。
(2) 乳液PCR/微珠富集
在微反应器中加入测序模板、PCR反应元件、微珠和引物,进行乳液PCR(Emulsi-on PCR)。PCR完成之后,变性模板,富集带有延伸模板的微珠,去除多余的微珠。微珠上的模板3’经过修饰,可以与玻片共价结合。乳液PCR最大的特点是可以形成数目庞大的独立反应空间以进行DNA扩增,其关键技术是“注水到油”,基本过程是在PCR反应前,将包含PCR所有反应成分的水溶液注入到高速旋转的矿物油表面,水溶液瞬间形成无数个被矿物油包裹的小水滴。这些小水滴就构成了独立的PCR反应空间。理想状态下,每个小水滴只含一个DNA模板和一个P1磁珠,由于水相中的P2引物和磁珠表面的P1引物所介导的PCR反应,这个DNA模板的拷贝数量呈指数级增加。PCR反应结束后,P1磁珠表面就固定有拷贝数目巨大的同来源DNA模板扩增产物。
(3) 微珠沉积
3’末端修饰的微珠沉积在一块玻片上,在微珠上样的过程中,沉积小室将每张玻片分成1个、4个或8个测序区域。SOLiD系统最大的优点就是每张玻片能容纳更高密度的微珠,在同一系统中轻松实现更高的通量。
(4) 连接测序
SOLiD连接反应的底物是8碱基单链荧光探针混合物,它的独特之处在于没有采用惯常的聚合酶,而用了连接酶。连接反应中,这些探针按照碱基互补规则与单链DNA模板链配对。探针的5’末端分别标记了CY5、Texas Red、CY3、6-FAM这4种颜色的荧光染料。探针3’端1~5位为随机碱基,可以是ATCG四种碱基中的任何一种碱基,其中第1、2位构成的碱基对是表征探针染料类型的编码区,双碱基编码矩阵规定了该编码区16种碱基对和4种探针颜色的对应关系,而3~5位的“n”表示随机碱基,6~8位的“z”指的是可以和任何碱基配对的特殊碱基。
(5) 数据分析
SOLiD使用连接法测序获得基于“双碱基编码原理”的颜色编码序列,随后系统自带的分析软件可先根据“双碱基编码矩阵”把reference碱基序列转换成色球序列,然后与SOLiD测序后所得色球序列进行比对,以此获取所测序列在reference的位置,及两者的匹配性信息。SOLiD系统自带的分析软件Life Scope可以完成上述从图像到mapping的分析过程。
SOLiD系统具有无可比拟的高通量,可拓展性、精确性和应用灵活性,在对复杂基因组进行研究的准确度高于目前现有的其它新一代测序技术。
2.技术特点
(1) SOLiD 4测序系统(图2-8)
A. 高通量:测序通量大于20GB/次,读长可达50个碱基(随机片段测序),50+35碱基(双端测序)和2×50个碱基(末端配对测序)。
B. 高准确度:连接反应、创新的双碱基编码技术和可选择性重复连接反应步骤保证了测序的高度准确性。
C. 易操作:样品制备支持随机片段文库和最高达10Kb插入片段的末端配对文库。无人工作模式,简单操作流程,流水线式自动数据分析,都使得该系统易于操作。
D. 灵活:两个独立控制的流动池,可以进行一张或二张玻片的同时分析。开放式高密度磁珠配置,每张玻片可分为1个、4个或8个测序区域,支持多重Barcode技术。
E. 应用广泛:支持大规模测序和结构变异性分析。独特的双碱基编码特性使系统具备自检功能,对SNP的检测精确率可以达到最高。以最大通量的标签实验为应用的基础,可以为全转录组、ChIP、microRNA和甲基化研究等提供高度敏感的检测方法。

图2-8. SOLiD 4 测序系统
(2) SOLiD 5500XL测序系统(图2-9)
建立于多年来精湛卓越的工程学设计传统的基础之上,全新的SOLiD 5500XL系列测序平台以高度的精确性、灵活性和易用性,使深度测序走向新的高度。
A. 准确性更高:更胜一筹的突变检测,超精确检测模块(ECC)可提供高达99.99%的精确性,多达98%的可定位碱基质量值高于45。
B. 人性化的样品处理和结果展示:自动、可重复、模式化的微珠制备,手工操作时间通常少于1小时;优化的样品制备试剂盒;全套方案帮助满足您的资源和研究需求。
C. 灵活可变,应用广泛。

图2-9. SOLiD 5500XL测序系统
3.应用领域
(1) 基因组测序以及重测序
(2) 基因组表达图谱分析
(3) 外显子组重测序
(4) DNA甲基化测序
(5) RNA和microRNA研究
(6) ChIP、SNP研究以及甲基化分析等
5.参考文献
1. 全集因组测序:
(1) The 1000 Genomes Project Consortium, A map of human genome variation from population-sc-ale sequencing.Nature. 2010;467(7319): 1061-73.
(2) Rubin CJ, et al.Whole-genome resequencing reveals loci under selection during chicken dome-stication.Nature. 2010;464(7288): 587-91.
2. 靶区域测序:
Vissers LE, et al.A de novo paradigm for mental retardation.Nat Genet. 2010;42(12):1109-12.
3. 染色体免疫共沉淀测序:
Tallack MR, et al.A global role for KLF1 in erythropoiesis revealed by ChIP-seq in primary erythr-oid cells.Genome Res. 2010;20(8):1052-63.
4. 甲基化测序分析:
Edwards JR, et al. Chromatin and sequence features that define the fine and gross structure of geno-mic methylation patterns.Genome Res. 2010;20(7):972-80.
5. 转录本测序:
(1) Ashton-Beaucage D, et al.The exon junction complex controls the splicing of MAPK and other long intron-containing transcripts in Drosophila.Cell. 2010;143(2):251-62.
(2) Heinig M, et al.A trans-acting locus regulates an anti-viral expression network and type 1 diabe-tes risk.Nature. 2010;467(7314):460-4.
(3) Kim TK, et al. Widespread transcription at neuronal activity-regulated enhancers.Nature. 2010;465(7295):182-7.
6. 小RNA测序分析:
Linsen SE, et al.Limitations and possibilities of small RNA digital gene expression profiling.Nat M-ethods. 2009;6(7):474-6.
7. 基因表达谱分析:
Hashimoto S, et al. High-resolution analysis of the 5'-end transcriptome using a next generation DN-A sequencer.PLoS One. 2009;4(1).
8. RNA结构测序:
(1) Kertesz M, et al.Genome-wide measurement of RNA secondary structure in yeast.Nature. 2010;467(7311):103-7.
(2) Tsai MC, et al. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 2010;329(5992):689-93.
9. 宏基因组学:
Tyler HL, et al. Confirmation of the sequence of 'CandidatusLiberibacterasiaticus' and assessment of microbial diversity in Huanglongbing-infected citrus phloem using a metagenomic approach. Mol Plant Microbe Interact. 2009;22(12):1624-34.
4.样品要求
(1) 基因组DNA:10-60μg纯化后DNA(fragment文库10μg最优,mate-pair文库20-60μg最优)。
(2) RNA:不少于10μg的纯化后RNA。
(3) microRNA:不少于10μg的纯化后RNA。
(4) 外显子捕获:不少于3μg的基因组DNA。