Sequenom MassArray 芯片平台
MassARRAY分子量阵列技术是Sequenom公司推出的世界上领先的基因分析工具,通过引物延伸或切割反应与灵敏、可靠的MALDI-TOF质谱技术结合,实现基因分型、DNA甲基化检测,为基因组学研究提供兼顾灵敏度和特异性服务的中高通量技术平台,是目前唯一采用质谱法进行直接检测的设备。Sequenom MassARRAY系统反应体系为非杂交依赖性,不需要各种标记物,实验设计灵活,更可实现高达40重反应,是目前市场上拥有最高性价比的产品。
1. 检测原理
Sequenom检测基于基质辅助激光解吸附电离飞行时间质谱(Matrix-Assisted Laser d-esorption/Ionization–Time Of Flight,MALDI-TOF)原理。质谱仪器(图2-34)主要由两部分组成:基质附助激光解吸电离离子源(MALDI)和飞行时间质量分析器(Time-of-Flight, TOF)。
Sequenom MassARRAY系统主要是利用基质辅助激光解吸电离飞行时间质谱 (MA-LDI-TOFMS)进行分析(图2-35),即PCR扩增产物或者预处理样本在延伸单碱基后,将制备的样品分析物与芯片基质共结晶,将该晶体放入质谱仪的真空管,而后用瞬时纳秒强激光激发,由于基质分子经辐射所吸收的能量,导致能量蓄积并迅速产热,从而使基质晶体升华,核酸分子就会解吸附并转变为亚稳态离子,产生的离子多为单电荷离子,这些单电荷离子在加速电场中获得相同的动能,进而在一非电场漂移区内按照其质荷比率得以分离,在真空小管中飞行到达检测器。MALDI产生的离子常用飞行时间检测器来检测,离子质量越小,就越快到达。理论上讲,只要飞行管的长度足够,TOF检测器可检测分子的质量数是没有上限的。MassARRAY SNP检测的质谱范围为5,000到8,500Dalton。MassARRAY检测过程大体可分为分子生物学反应,质谱阵列的点样和质谱检测三个环节。
基于MassARRAY分子量阵列平台的iPLEX GOLD技术可以设计最高多达40重PCR反应和基因型检测,实验设计非常灵活,分型结果准确性高。根据应用需要,对于数十到数百个SNP位点进行数百至数千份样本检测时,MassARRAY具有最佳性价比。特别适合于对全基因组研究发现的结果进行验证,或者是有限数量的研究位点已经确定的情况。其中DNA甲基化分析采用MassARRAY EpiTYPER流程,该过程结合碱基特异性酶切(分子切割)和MALDI-TOF检测原理,可实现多重CpG的分析检测,同时具有DNA甲基化定量分析能力。Sequenom DNA甲基化检测由亚硫酸盐处理待测DNA开始,经过亚硫酸盐处理,DNA中未甲基化胞嘧啶(C)转变为尿嘧啶(U),而甲基化修饰的胞嘧啶不受影响,因此亚硫酸盐处理将产生甲基化特异的序列变化。通过PCR扩增过程引入T7-启动子序列,经T7 DNA聚合酶的体外转录过程得到各样本RNA产物,再经过碱基特异性酶切处理,得到RNA小片段,并用飞行质谱检测每个片段的分子量,最后EpiTYPER程序完成数据的自动化处理并报告每个检测片段的甲基化程度。
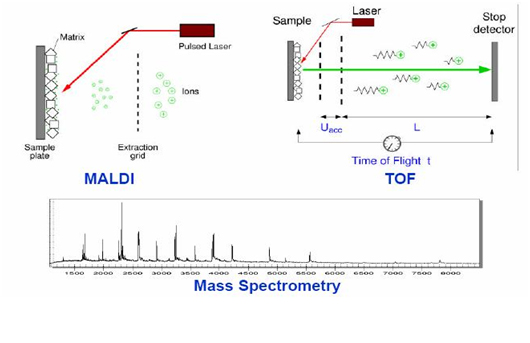
图2-33. Sequenom MassArray系统检测原理
2. 技术特点
(1) 高通量:一张芯片可对384个样本进行多重检测,每个体系最多可实现40重反应,而且通量可根据客户要求进行个性化调整。
(2) 高性价比:无需荧光标记,仅需合成普通引物,大大降低成本,单个分析成本低;适用范围广,可同时检测几十到成千上万个样本,检测几十到成百上千个位点。
(3) 高灵敏度:分析所需样本量少,且检测精度高,并能进行定量分析。
(4) 高灵活度:功能多样,适用于SNP分型以及DNA甲基化定量分析。而且一张芯片上样本数量和位置可随意选择,同时,一张芯片上样本和位点检测匹配也可随意选择。
3. 应用领域
(1) SNP分型质控
(2) SNP位点等位基因频率计算
(3) 体细胞突变检测质控和验证
(4) 甲基化定量分析
(5) 基因表达定量分析
(6) CNV检测分析
4. 参考文献
(1) Morelli G, et al.Yersinia pestis genome sequencing identifies patterns of global phylogenetic diversity.Nat Genet. 2010;42(12):1140-3.
(2) Oeth P, et al. Qualitative and quantitative genotyping using single base primer extension coupled with matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MassARRAY).Methods Mol Biol. 2009;578:307-43.
(3) Zheng W, et al. Genome-wide association study identifies a new breast cancer susceptibility locus at 6q25.1. Nat Genet. 2009;41(3):324-8.
(4) Sinotte M, et al. Genetic polymorphisms of the vitamin D binding protein and plasma concentrations of 25-hydroxyvitamin D in premenopausal women.Am J ClinNutr. 2009;89(2):634-40.
(5) Sun T, et al. A six-nucleotide insertion-deletion polymorphism in the CASP8 promoter is associated with susceptibility to multiple cancers.Nat Genet. 2007;39(5):605-13.
(6) Starinsky S, et al. Genotype phenotype correlations in Israeli colorectal cancer patients.Int J Cancer. 2005;114(1):58-73.
(7) Jurinke C, etal. Automated genotyping using the DNA MassArray technology. Methods Mol Biol. 2002;187:179-92.
(8) Jurinke C, et al. The use of MassARRAY technology for high throughput genotyping.AdvBiochemEngBiotechnol. 2002;77:57-74.
(9) Cui H, et al.Loss of imprinting in normal tissue of colorectal cancer patients with microsatellite instability. Nat Med. 1998;4(11):1276-80.